上田 泰己先生HIROKI UEDA
【現在の所属】
東京大学大学院 医学系研究科 システムズ薬理学教室 教授
理化学研究所 生命機能科学研究センター 合成生物学研究チーム チームリーダー
【受賞当時の所属】
理化学研究所 発生・再生科学総合研究センター
システムバイオロジー研究チーム チームリーダー
1975年福岡県生まれ。2000年東京大学医学部卒業、2004年同大大学院医学系研究科修了。 2003年から理化学研究所にてチームリーダー、2009年からプロジェクトリーダー、2011年からグループディレクターを経て2013年より東京大学大学院医学系研究科教授。現在、理化学研究所チームリーダー、東京大学大学院情報理工学研究科教授などを兼務。「細胞を創る」研究会会長(2008年)。
睡眠・覚醒リズムのリン酸化仮説
2019.12.16
「フロンティアサロンで講演してくれませんか?」。それは東京農工大の大野弘幸学長からの一本の電話から始まりました。2010年9月13日の午後5時から銀座の交詢社倶楽部に伺い、株式会社ナガセの永瀬昭幸社長、東京農工大の宮田清藏元学長、JSTの故北澤宏一理事長をはじめとする各分野の錚々たる先生方の前で「時間の生命科学」と題して研究の発表をさせていただきました。発表のハイライトは、体内時計の温度補償性の解明に関して。体内時計の温度補償性とは、温度が高いときも温度が低いときも地球の自転周期である24時間にほぼ近い周期で発振する性質です。この温度補償性は、私達の体内時計が地球の自転を模すために満たすべき必然の性質である一方で(※暑い赤道でも寒い北極・南極でも地球の自転周期は変わりません)、温度が10℃高くなると酵素反応の速度は2倍から3倍程度早くなるという生化学の常識からは大きく逸脱した不思議な性質でもあり、1960年に発見されてから長い間謎とされていました。私達は、前年の2009年に世界で初めて、哺乳類の体内時計の周期決定に中心的な役割を果たしているリン酸化酵素が、その酵素反応速度が温度に全く依存しない性質をもっていることを突き止めました。その発見の説明を終えた時です。「すると時間は濃度なのか?」と北澤先生から鋭い質問を頂きました。私がとっさに考えて出した回答は「おそらく違います。時間はタンパク質の構造に刻み込まれるものだと思います」。以来、生命の時間に関する北澤先生の質問の答えを探し続けています。果たして生体内の時間は「濃度」のように分子の「量」として細胞内に溜まっていくものとして表現されているものなのでしょうか。それとも「構造」として、例えば分子のリン酸化状態などのように、分子の「質」に刻み込まれていくものなのでしょうか。
分子のリン酸化修飾は細胞内のタンパク質機能調整において中心的な役割を果たすことが知られています。しかし、リン酸化修飾によって生じるタンパク質1分子の「質」的な活性変化が個体全体の表現型に直結して表出する例は稀です。概日時計研究はその稀有な例であり、当初はタンパク質の活性修飾を行う一要素と捉えられていたリン酸化修飾が、実際は細胞の表現型を支配していることを明らかにしてきました。これまでに私達は哺乳類においてリン酸化制御が遺伝子の発現やタンパク質分解の量的変動とは独立して個体全体の行動リズムを支配し(Ode et al, 2017)、精製基質と酵素から再構成されるリン酸化活性に、概日時計の24時間制御の中核となる性質(温度補償性)が保たれることを示してきました(Isojima et al, 2009)。私達は2017年にこの温度「非」感受性のリン酸化酵素内に温度センサー部位があり、温度が高くなると酵素反応を遅くすることで、リン酸化酵素反応のスピードが温度によって変化しないように調節している仕組みがあることを解明しました。大変面白いことに、この部位を他の温度感受性のリン酸化酵素に移植すると温度「非」感受性に変化することを示しました(Shinohara et al, 2017)。
それではリン酸化による分子の質的変動は、個体全体の時間情報の担い手として、どこまで一般化可能なのでしょうか。高次脳機能の基盤ともなる睡眠・覚醒リズムは、一見して概日時計発振よりも複雑な生理機能です。フロンティアサロンで講演した2010年頃から私達は睡眠・覚醒リズムの研究をモデルにしながら個体の時間を紐解く研究を開始しました。私達は、分子活性の変動そのものが睡眠覚醒リズムの時間情報、特に睡眠時間長を支配する可能性を考え、哺乳類の睡眠(特にノンレム睡眠)について、神経細胞の活動パターンを直接担うイオンチャネル・ポンプについて、ノンレム睡眠時の神経活動の数理モデリングと、マウス睡眠表現型のスクリーニングシステム(SSS法)、独自に改良したCRISPRを用いたノックアウトマウス作製技術(Triple-CRISPR法)を用いて(図1)、細胞内Ca2+動態に直接関与する一連のイオンチャネル・ポンプが睡眠時間制御に重要であることを見出してきました(Sunagawa et al, 2016, Tatsuki et al, 2016)。さらに、細胞内Ca2+が制御するリン酸化酵素に着目し、Camk2a/bノックアウトマウスが著明な睡眠時間の短縮を示すことを明らかにしています(Tatsuki et al, 2016)。このことはCaMKIIα/βが睡眠を誘導するリン酸化酵素であることを意味し、睡眠研究における史上初の睡眠誘導性リン酸化酵素の発見となりました。発表以後、2016年および翌年の2017には他グループによって睡眠制御因子として異なるリン酸化酵素(いずれも睡眠誘導性)が報告されています。私達は、睡眠誘導性リン酸化酵素CaMKIIα/βの発見を元に、神経細胞の興奮持続やエネルギーの枯渇、外的環境変化によるストレスなどの細胞状態・個体状態の履歴をリン酸化を中心とした分子修飾として統合・記録し、神経細胞の興奮性の低下、代謝活動の制御、ストレスによる細胞障害の修復を誘導する睡眠の「リン酸化仮説」を提唱するに至りました(Tatsuki et al, 2016, 図2)。私たちの概日時計研究や睡眠・覚醒リズムの研究からも示されるように、リン酸化制御が数分から数時間の広いスケールの時間情報の本体となりうることが示されつつあります。フロンティアサロンで出会った質問から9年以上がたちました。細胞の中の概日時計の仕組みだけではなく、個体の中の睡眠・覚醒リズムの仕組みからもその答えがまさに得られつつあります。これらの科学的発見やその過程で成した技術的発明が私達をどこまで連れて行ってくれるか大変楽しみです。
References:
1.Isojima et al, PNAS, 106(37):15744-9 (2009), 2. Susaki et al. Cell, 157(3): 726–39, (2014), 3. Tainaka et al. Cell, 159(6):911-24(2014), 4. Sunagawa et al, Cell Reports, 14(3):662-77 (2016), 5. Ode et al, Molecular Cell, 65, 176–190 (2017), 6. Shinohara et al, Molecular Cell. 67, 783-798 (2017), 7. Ukai et al, Nat. Protoc. 12, 2513-2530 (2017), 8. Niwa et al, Cell report, 24, 2231–2247 (2018).
図1.睡眠・覚醒リズムのシステム生物学研究を支える3つの基盤技術
タンパク質のリン酸化と睡眠覚醒の時間情報処理の因果関係を明らかにするため、遺伝子ノックアウト・ノックインマウスの高速に作出し、その(睡眠)表現型解析を並列的に行う次世代遺伝学技術を開発した(図1A)。ここで次世代遺伝学は、交配を必要としない遺伝学という意味で用いる。次世代遺伝学を実現するためにまず、これまでにマウス受精卵やES細胞に3つのガイドRNAとCRISPR/Cas9を導入することで、ほぼ全身の細胞について両アレルの遺伝子がノックアウトされたマウスを交配なしに一世代で得るTriple-CRISPR法を開発している(図1B左)。また、遺伝子改変されたES細胞から、このES細胞にほぼ全身の細胞が由来するESマウス法を確立し、遺伝子ノックインマウスの並列作製にも成功している(図1B右)。これらの遺伝子改変マウスの睡眠表現型は、呼吸波形を元にした睡眠覚醒状態の判定法であるSSS法を用いて解析する(図1D、E)。我々はこれらの手法を用いて、~1,000匹/週のマウスの睡眠表現型解析が可能なシステムを恒常的に運用している。SSS法では、人工知能を用いた改良が進展しており、NREM/REM睡眠の差異も呼吸波形から自動判定が可能となりつつある(図1E)。ノックイン・ノックアウトマウスの個体表現型が、どの脳領域・神経回路の活性変動に対応するのかを1細胞レベルで検証することも可能である。我々はマウスの全脳および全身の組織透明化・1細胞解像度イメージングに成功している(CUBIC法)(図1C)。特にc-Fosに対する免疫染色を用いて神経活動情報を取得し多数の神経細胞マーカータンパク質に対する3次元免疫染色像との比較解析を行うことで場所を特定し、さらに部位特異的な変異体の発現や遺伝子のノックアウトを行うことで、生体時間の細胞レベルでの機構の解明に迫ることが可能である。これらの3つの基盤技術はすでに恒常的に運用されている。Triple-CRISPR(図1A, B)およびES-mouse技術(図1A, B)により、それぞれ遺伝子ノックアウトマウス・ノックインマウスが作出され、交配を経ることなく直接表現型解析が可能である。睡眠表現型は、東大ではおよそ216匹/週が、理研では936匹/週が測定され(図1D,E)、測定結果は自動的に95%以上の精度でもってデータ解析が為される。これは、世界最大のスループットをもった、マウス睡眠表現型の解析施設である。また、タンパク質発現状況の詳細は、一連のCUBIC法を用いて全脳全細胞について一細胞解像度で比較解析が可能であり結果を速やかに共有する体制も構築されている(図1C)。

図1.個体システム生物学を支える3つの基盤技術
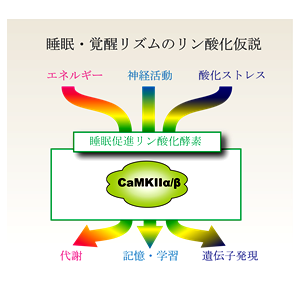
図2.睡眠・覚醒リズムのリン酸化仮説
2016年に提唱した睡眠・覚醒リズムのリン酸化仮説(Tatsuki et al, 2016)では、覚醒中に起きる神経活動をはじめとした様々な細胞内の活動によって睡眠促進リン酸化酵素が活性化され、睡眠状態を誘導することを提唱しています。
(1)覚醒中のどのような細胞内の活動が睡眠促進リン酸化酵素を活性化するのか?(眠気や疲れは何によって書き込まれるのか)
(2)睡眠促進リン酸化酵素が活性化状態はどのような形で保持されているのか?(眠気や疲れはどのような記憶されるのか?)
(3)睡眠中の様々な細胞内の活動の変化が引き起こされるのか?((眠気や疲れがどのように細胞状態の変化を引き起こすのか?)
などの仕組みの解明が待たれます。CaMKIIα/βの場合は、(1)カルシウムによってCaMKIIα/βの活性化が引き起こされ、(2)活性化状態は、CaMKIIα/βの自己リン酸化状態として保持され、(3)CaMKIIα/βのリン酸化酵素活性によって下流の細胞状態の変化が引き起こされる、ことが想定されます。